Python 使用TensorFlow 进行皮肤癌检测
早期检测任何疾病,尤其是癌症,对治疗阶段非常关键。在这方面做出的一项努力就是使用机器学习算法来检测和诊断皮肤癌,借助于像 TensorFlow 这样的机器学习框架。
传统的癌症检测方法非常耗时,并且需要专业的皮肤科医生。然而,借助于 TensorFlow,不仅可以加快这个过程,而且更准确和高效。此外,那些不能及时获得医生和皮肤科医生的人也可以在此期间使用它。
步骤
步骤1 - 导入像 numpy、pandas、matplotlib 和 seaborn 等库,并加载图像数据集并将其存储为列表。
步骤2 - 将图像列表加载为 pandas 数据帧,并提取列表中每个图像的两个标签。
步骤3 - 将标签转换为符号 0 和 1,以简化比较每个标签下的图像数量,并使用饼图进行可视化。
步骤4 - 如果不存在不平衡,打印每个标签的一些图像。
步骤5 - 将数据集划分为训练集和测试集。
步骤6 - 创建用于图像输入的流水线。
步骤7 - 使用 EfficientNet 架构创建和编译模型。
步骤8 - 训练模型至少5个周期。
步骤9 - 可视化训练损失和验证损失之间的差异。
示例
在这个示例中,我们将使用包含两种类型图像的皮肤癌数据集来开发一个模型,您可以在此处找到。然后,我们将借助于 TensorFlow 来开发一个模型,以便在没有太多训练的情况下获得期望的结果。为此,我们还将使用 EfficientNet 架构获取预训练的权重。
#import the required libraries
import numpy as np
import pandas as pd
import seaborn as sb
import matplotlib.pyplot as plt
from glob import glob
from PIL import Image
from sklearn.model_selection import train_test_split
import tensorflow as tf
from tensorflow import keras
from keras import layers
from functools import partial
AUTO = tf.data.experimental.AUTOTUNE
import warnings
warnings.filterwarnings('ignore')
#load the dataset
images = glob('train/*/*.jpg')
len(images)
#create dataset and extract labels
images = [path.replace('', '/') for path in images]
df = pd.DataFrame({'filepath': images})
df['label'] = df['filepath'].str.split('/', expand=True)[1]
print(df.head())
df['label_bin'] = np.where(df['label'].values == 'malignant', 1, 0)
df.head()
#check if both types of files are same in number
x = df['label'].value_counts()
plt.pie(x.values,
labels=x.index,
autopct='%1.1f%%')
plt.show()
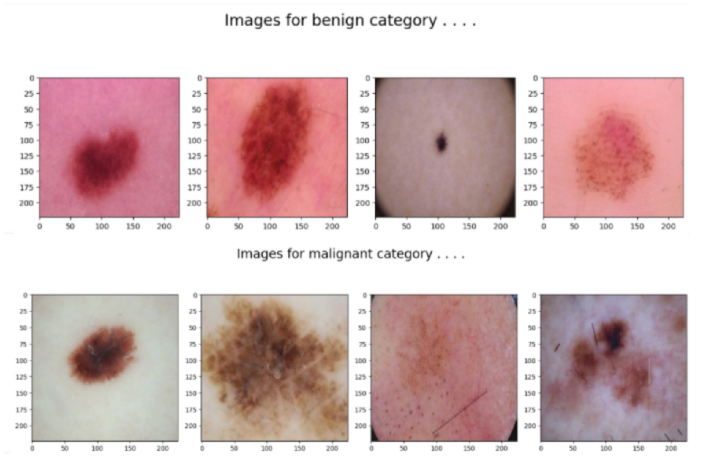
#printing the images of the two categories
for cat in df['label'].unique():
temp = df[df['label'] == cat]
index_list = temp.index
fig, ax = plt.subplots(1, 4, figsize=(15, 5))
fig.suptitle(f'Images for {cat} category . . . .', fontsize=20)
for i in range(4):
index = np.random.randint(0, len(index_list))
index = index_list[index]
data = df.iloc[index]
image_path = data[0]
img = np.array(Image.open(image_path))
ax[i].imshow(img)
plt.tight_layout()
plt.show()
#split the dataset into train and test
features = df['filepath']
target = df['label_bin']
X_train, X_val,\
Y_train, Y_val = train_test_split(features, target,
test_size=0.15,
random_state=10)
X_train.shape, X_val.shape
def decode_image(filepath, label=None):
img = tf.io.read_file(filepath)
img = tf.image.decode_jpeg(img)
img = tf.image.resize(img, [224, 224])
img = tf.cast(img, tf.float32) / 255.0
if label == None:
return img
return img, label
#create pipelines for image input
train_ds = (
tf.data.Dataset
.from_tensor_slices((X_train, Y_train))
.map(decode_image, num_parallel_calls=AUTO)
.batch(32)
.prefetch(AUTO)
)
val_ds = (
tf.data.Dataset
.from_tensor_slices((X_val, Y_val))
.map(decode_image, num_parallel_calls=AUTO)
.batch(32)
.prefetch(AUTO)
)
#building the model architecture using Keras API
from tensorflow.keras.applications.efficientnet import EfficientNetB7
pre_trained_model = EfficientNetB7(
input_shape=(224, 224, 3),
weights='imagenet',
include_top=False
)
for layer in pre_trained_model.layers:
layer.trainable = False
from tensorflow.keras import Model
inputs = layers.Input(shape=(224, 224, 3))
x = layers.Flatten()(inputs)
x = layers.Dense(256, activation='relu')(x)
x = layers.BatchNormalization()(x)
x = layers.Dense(256, activation='relu')(x)
x = layers.Dropout(0.3)(x)
x = layers.BatchNormalization()(x)
outputs = layers.Dense(1, activation='sigmoid')(x)
model = Model(inputs, outputs)
model.compile(
loss=tf.keras.losses.BinaryCrossentropy(from_logits=True),
optimizer='adam',
metrics=['AUC']
)
#train the model for 5 epochs
history = model.fit(train_ds,
validation_data=val_ds,
epochs=5,
verbose=1)
#checking the loss
hist_df = pd.DataFrame(history.history)
hist_df.head()
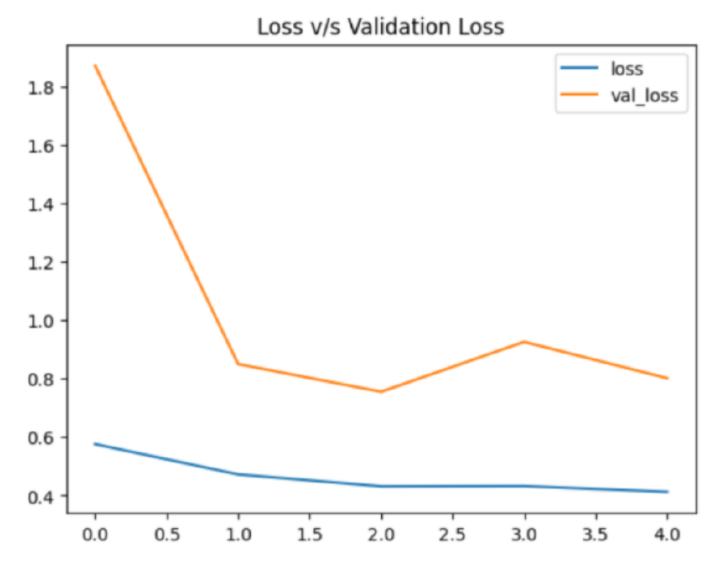
#plotting line graph
hist_df['loss'].plot()
hist_df['val_loss'].plot()
plt.title('Loss v/s Validation Loss')
plt.legend()
plt.show()
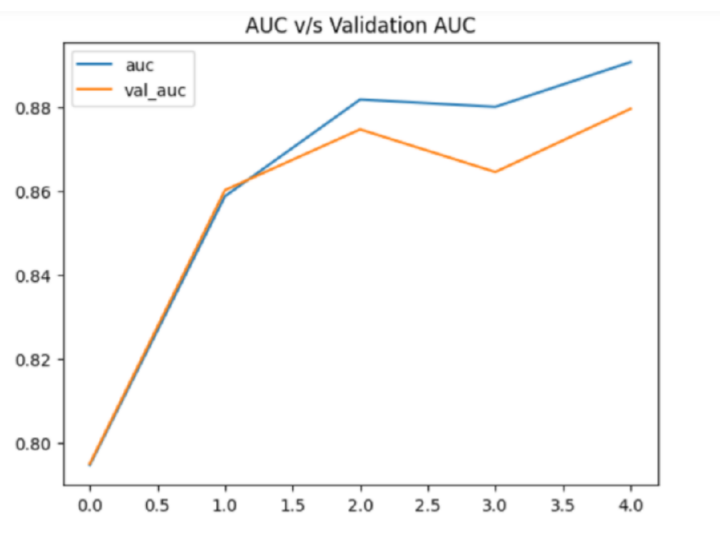
hist_df['auc'].plot()
hist_df['val_auc'].plot()
plt.title('AUC v/s Validation AUC')
plt.legend()
plt.show()
我们首先加载存储在本地系统中的图像,然后创建一个数据帧来存储所有的文件路径和加载的标签。存储的标签被转换为二进制格式,恶性标签表示为1,其他标签表示为0。 代码的后半部分绘制了一个饼图,可视化了标签类别的分布,并计算了每个类别的出现次数。 然后,我们从每个类别中随机选择4个图像,并使用Matplotlib以1×4的网格打印它们。decode_image()函数读取图像文件,对图像进行解码和调整大小。然后使用fit()方法训练模型,并进行训练。然后使用fit()方法返回的history对象提取训练和验证损失。然后将这些值存储在一个数据帧中。 使用Python的Matplotlib库绘制损失和验证损失值。 输出
filepath label
0 train/benign/100.jpg benign
1 train/benign/1000.jpg benign
2 train/benign/1001.jpg benign
3 train/benign/1002.jpg benign
4 train/benign/1004.jpg benign
第1个周期/共5个周期
71/71 [==============================] - 28s 356ms/步 - 损失: 0.5760 - AUC: 0.7948 - 验证损失: 1.8715 - 验证AUC: 0.7951
第2个周期/共5个周期
71/71 [==============================] - 25s 348ms/步 - 损失: 0.4722 - AUC: 0.8587 - 验证损失: 0.8500 - 验证AUC: 0.8602
第3个周期/共5个周期
71/71 [==============================] - 24s 336ms/步 - 损失: 0.4316 - AUC: 0.8818 - 验证损失: 0.7553 - 验证AUC: 0.8746
第4个周期/共5个周期
71/71 [==============================] - 24s 331ms/步 - 损失: 0.4324 - AUC: 0.8800 - 验证损失: 0.9261 - 验证AUC: 0.8645
第5个周期/共5个周期
71/71 [==============================] - 24s 344ms/步 - 损失: 0.4126 - AUC: 0.8907 - 验证损失: 0.8017 - 验证AUC: 0.8795
结论
虽然TensorFlow在皮肤癌检测模型中表现良好,但它也有一些缺点,比如使用大量计算资源或大量内存等。因此,尝试其他框架如PyTorch、Keras和MXNet等对于使用机器学习进行皮肤癌检测领域的更多可能性值得一试。
 极客笔记
极客笔记