Python 使用PCA降低数据维度
在机器学习算法中使用的任何数据集都可能具有多个维度。然而,并非所有维度都对给出高效输出有所贡献,只会因为尺寸和复杂性增加而导致机器学习模型表现不佳。因此,消除数据集中的这些特征变得十分重要。为此,我们使用一种称为PCA的降维算法。
主成分分析(PCA)可帮助从数据集中删除不对优化结果有帮助的维度,从而创建一个较小且较简单的数据集,其中包含原始和有用信息的大部分内容。PCA基于特征提取的概念,即当将高维空间中的数据映射到低维空间时,后者的方差应该最大。
语法
pca = PCA(n_components = number)
在这里,PCA是执行降维的类,pca是从中创建的对象。它只需要一个参数 – 我们想要的主成分的数量。
此外,当与fit()、DataFrame()和head()函数一起使用时,它会返回包含主成分的新数据集作为表格,如我们在示例中所见。
步骤
- 第1步 - 导入Python的sklearn和pandas库,以及相关的子模块。
-
第2步 - 加载所需的数据集并将其转换为pandas数据帧。
-
第3步 - 使用标准缩放器对特征进行标准化,并将新数据集存储为pandas数据帧。
-
第4步 - 在缩放后的数据集上使用PCA类并创建一个对象。同时,传递主成分的数量,并根据需要适应并显示结果数据。
示例1
在这个例子中,我们将使用Python的sklearn库中已经存在的load_diabetes数据集来演示PCA的应用。为了做到这一点,我们将使用PCA类,但在此之前,我们需要处理和标准化数据集。
#import the required libraries
from sklearn import datasets #to get the load_diabetes dataset
from sklearn.preprocessing import StandardScaler #this standardizes the dimensions
from sklearn.decomposition import PCA #to perform PCA
from sklearn.datasets import load_diabetes #the dataset that we will use to perform PCA
import pandas as pd #to work with the dataframes
#load the dataset as pandas dataframe
diabetes = datasets.load_diabetes()
df = pd.DataFrame(diabetes['data'], columns = diabetes['feature_names'])
df.head() #displays the data frame when run in a different cell
#standardize the dimensions by creating the object of StandardScaler
scalar = StandardScaler()
scaled_data = pd.DataFrame(scalar.fit_transform(df))
scaled_data #displays the dataframe after standardization when run in a different cell
#apply pca
pca = PCA(n_components = 4)
pca.fit(scaled_data)
data_pca = pca.transform(scaled_data)
data_pca = pd.DataFrame(data_pca,columns=['PC1','PC2','PC3', 'PC4'])
data_pca.head()
加载糖尿病数据,返回一个类似于字典的对象,从中提取数据到Pandas Dataframe中。我们使用StandardScaler对数据进行标准化,并应用fit_transform()方法到创建的Pandas Dataframe。
在标准化的数据上调用fit()方法,将进行PCA分析。分析结果的数据再次存储在一个单独的数据帧中,并按如下所示打印出来。
输出

由于我们选择了4个主成分,所以返回的输出有4列分别表示每个主成分。
示例2
在这个示例中,我们将从sklearn库中取得load_wine数据集。同时,这次我们将主成分的数量设置为3。
#import the required libraries
from sklearn import datasets #to get the load_wine dataset
from sklearn.preprocessing import StandardScaler #this standardizes the dimensions
from sklearn.decomposition import PCA #to perform PCA
from sklearn.datasets import load_wine #the dataset that we will use to perform PCA
import pandas as pd #to work with the dataframes
#load the dataset as pandas dataframe
wine = datasets.load_wine()
df = pd.DataFrame(wine['data'], columns = wine['feature_names'])
df.head() #displays the dataframe when run in a different cell
#standardize the dimensions by creating the object of StandardScaler
scalar = StandardScaler()
scaled_data = pd.DataFrame(scalar.fit_transform(df))
scaled_data #displays the dataframe after standardization when run in a different cell
#apply pca
pca = PCA(n_components = 3)
pca.fit(scaled_data)
data_pca = pca.transform(scaled_data)
data_pca = pd.DataFrame(data_pca,columns=['PC1','PC2','PC3'])
data_pca.head()
输出
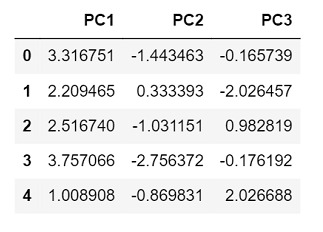
由于我们这次只选择了3个主成分,所以返回的输出只有3列,分别表示每个主成分。
结论
PCA不仅使数据集的各个成分相互独立,还通过减少数据集的特征数量来解决过拟合的问题。然而,降维不仅限于PCA。还有其他方法,如LDA – 线性判别分析和GDA – 广义判别分析,可以实现相同的效果。
 极客笔记
极客笔记