Python中的ANOVA测试
以下教程基于数据分析;我们将详细讨论 方差分析(ANOVA) ,以及在Python编程语言中进行方差分析的过程。ANOVA通常在 心理学 研究中使用。
在接下来的教程中,我们将了解如何利用 SciPy 库来进行ANOVA,并在Python中使用 Pyyttbl 和 Statsmodels 进行 手动 评估。
了解ANOVA测试
我们可以将 方差分析 测试,也称为 ANOVA ,视为对多个组进行T检验的概括。通常情况下,我们使用独立的T检验来比较两组状态的均值。当我们需要比较两组以上状态的均值时,我们使用ANOVA测试。
ANOVA测试检查模型中的平均值是否存在差异(检查是否存在整体效应);然而,该方法并不告诉我们差异的位置(如果有差异的话)。我们可以通过进行事后检验来找出组之间的差异位置。
然而,为了进行任何测试,我们首先必须定义零假设和备择假设:
- 零假设: 组之间没有显著差异。
- 备择假设: 组之间存在显著差异。
我们可以通过比较两种变异来进行ANOVA测试。第一种变异是样本均值之间的变异,另一种是每个样本内的变异。下面显示的公式描述了单因素ANOVA测试统计量。
ANOVA公式的输出 F统计量 (也称为 F比率 ),可以用于分析多组数据以确定样本之间和样本内的变异性。
我们可以将单因素ANOVA测试的公式写成如下所示:

其中,
y i - 第 **i th ** 组的样本平均值
n i - 第 **i th ** 组的观察值数量
y – 数据的总平均值
k – 组的总数量
y ij - 第 **j th ** 个组之外的观察值
N – 总样本量
当我们制作ANOVA表时,可以以以下格式看到上述所有组成部分:
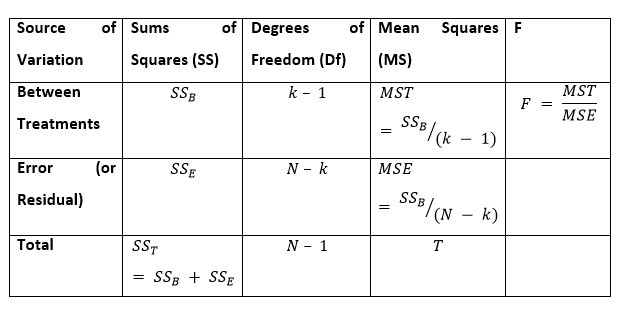
通常,如果F值对应的p值小于0.05,则可以排除零假设,维持备择假设。在零假设被拒绝的情况下,我们可以说所有组的均值不相等。
注意:如果被测试的组之间不存在真实差异,也就是被称为零假设,ANOVA测试的F比值将接近于1。
ANOVA测试的假设条件
在进行ANOVA测试之前,我们必须满足以下假设条件,如下所示:
- 我们可以随机独立地从由因素水平定义的总体中获取观察值。
- 每个因素水平的数据是普遍分布的。
- 独立性: 样本个案之间必须是相互独立的。
- 方差齐性: 齐性意味着组之间的方差大致相等。
我们可以通过类似Brown-Forsythe测试或Levene测试的帮助来测试方差齐性的假设。我们还可以通过直方图、峰度或偏度值,或者通过Kolmogorov-Smirnov、Shapiro-Wilk或Q-Q图等测试来测试评分分布的正态性。我们还可以从研究设计中确定独立性的假设。
值得注意的是,ANOVA测试在违反独立性假设时不具有鲁棒性。这意味着即使有人试图违反正态性或齐性的假设,他们仍然可以进行测试并信任结果。
然而,如果违反了独立性假设,ANOVA测试的结果是不可接受的。通常情况下,在我们有均等大小的组的情况下,即使存在齐性的违规,分析也被认为具有鲁棒性。如果样本量较大,即使存在正态性的违规,重新进行ANOVA测试通常也是可以接受的。
了解ANOVA测试的类型
ANOVA测试可以分为三种主要类型。这些类型如下所示:
- 单因素ANOVA测试
- 双因素ANOVA测试
- n因素ANOVA测试
单因素ANOVA测试
只有一个独立变量的方差分析测试被称为 单因素ANOVA测试 。
例如,一个国家可以评估冠状病毒病例的差异,而一个国家可以有多个用于比较的类别。
双因素ANOVA测试
具有两个独立变量的方差分析测试被称为 双因素ANOVA测试 。这个测试也被称为 因子方差分析测试 。
例如,扩展上面的示例,双向ANOVA可以考察 冠状病毒 (因变量)在 年龄组 (第一个自变量)和 性别 (第二个自变量)之间的差异。双向ANOVA可用于检查这两个自变量之间的交互作用。交互作用意味着差异在自变量的所有类别中不均匀存在。
假设 老年组 的冠状病毒病例总体上可能比 年轻组 更多;然而,这种差异可能在 欧洲 国家与 亚洲 国家之间存在差异。
n维ANOVA测试
如果研究人员使用了两个以上的自变量,那么方差分析(ANOVA)测试就被称为 n维ANOVA测试 。这里的n代表我们拥有的自变量数量。该测试也被称为 多元方差分析(MANOVA)测试 。
例如,我们可以同时使用诸如国家、年龄组、性别、种族等多个自变量来研究 冠状病毒 的潜在差异。
ANOVA测试将提供我们一个单一的(单变量)F值;然而,MANOVA测试将提供我们一个多元的F值。
理解ANOVA中的重复和非重复设计
通常,我们可能会听到关于ANOVA测试中的重复和非重复设计。让我们了解一下它们的含义:
带重复设计的双向ANOVA测试
带有重复设计的双向ANOVA测试是在两个组中进行多个任务操作的情况下进行的。
例如,假设冠状病毒疫苗仍在研发阶段。医生们正在对受感染的两组患者进行两种不同的治疗。
不带重复设计的双向ANOVA测试
在只有一个组的情况下进行不带重复设计的双向ANOVA测试,我们会对同一组进行两次测试。
例如,假设疫苗已成功研发,并且研究人员正在对一组志愿者进行疫苗接种前后的测试,以观察疫苗是否有效。
理解ANOVA后续测试
在进行ANOVA测试时,我们试图确定群体之间是否存在统计显著差异。如果存在,我们将需要测试差异的具体位置。
因此,研究人员使用事后检验来检查哪些群体之间存在差异。
我们可以进行事后检验,即t检验,检查群体之间的均值差异。我们可以进行多个多重比较测试以控制第一类错误率,包括Bonferroni、Dunnet、Scheffe和Turkey测试。
现在,我们将使用Python编程语言来解析仅涉及一元ANOVA测试。
使用Python理解一元ANOVA测试
我们将执行ANOVA测试的过程分为不同的部分。
导入所需的库
为了开始使用ANOVA测试,让我们导入一些项目所需的必要库和模块。
语法:
import pandas as pd
import matplotlib.pyplot as plt
import statsmodels.api as sm
from statsmodels.formula.api import ols
import seaborn as sns
import numpy as np
import pandas.tseries
plt.style.use('fivethirtyeight')
假设
让我们考虑一个关于问题的假设:
“对于每一种饮食,人们的体重平均值是相同的。”
加载数据
在下面的问题中,我们将使用由谢菲尔德大学设计的一个饮食数据集。该数据集包含一个性别的二元变量,其中1表示男性,0表示女性。
让我们考虑以下相同的语法:
语法:
mydata = pd.read_csv('Diet_Dataset.csv')
了解数据集
成功导入数据集后,让我们打印一些数据以了解其情况。
示例
print(mydata.head())
输出:
Person gender Age Height pre.weight Diet weight6weeks
0 25 41 171 60 2 60.0
1 26 32 174 103 2 103.0
2 1 0 22 159 58 1 54.2
3 2 0 46 192 60 1 54.0
4 3 0 55 170 64 1 63.3
现在让我们打印出数据集中的总行数。
示例
print('The total number of rows in the dataset:', mydata.size)
输出:
The total number of rows in the dataset: 546
检查缺失值
现在,我们需要查看数据集中是否有任何缺失的值。我们可以使用以下语法来进行检查。
示例
print(mydata.gender.unique())
# displaying the person(s) having missing value in gender column
print(mydata[mydata.gender == ' '])
输出:
[' ' '0' '1']
Person gender Age Height pre.weight Diet weight6weeks
0 25 41 171 60 2 60.0
1 26 32 174 103 2 103.0
我们可以观察到’gender’列中有两个条目包含缺失值。现在让我们计算数据集中缺失值的总百分比。
示例
print('Percentage of missing values in the dataset: {:.2f}%'.format(mydata[mydata.gender == ' '].size / mydata.size * 100))
输出:
Percentage of missing values in the dataset: 2.56%
如我们所见,在数据集中大约有3%的缺失值。我们可以选择忽略、删除或者根据最接近的身高均值来分类性别。
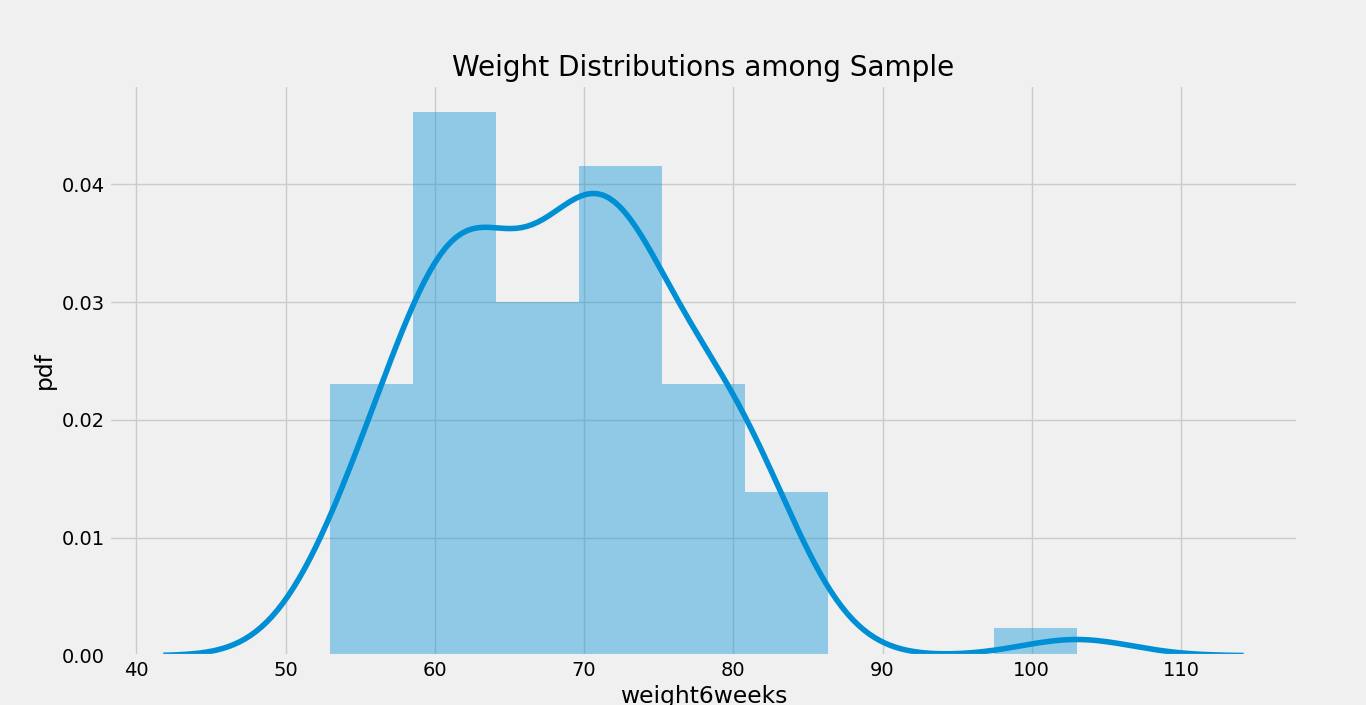
理解体重的分布
在接下来的步骤中,我们将使用 distplot() 函数绘制一个图表,以了解样本数据中体重的分布情况。让我们考虑以下的代码片段。
示例
f, ax = plt.subplots( figsize = (11,9) )
plt.title( 'Weight Distributions among Sample' )
plt.ylabel( 'pdf' )
sns.distplot( mydata.weight6weeks )
plt.show()
输出:
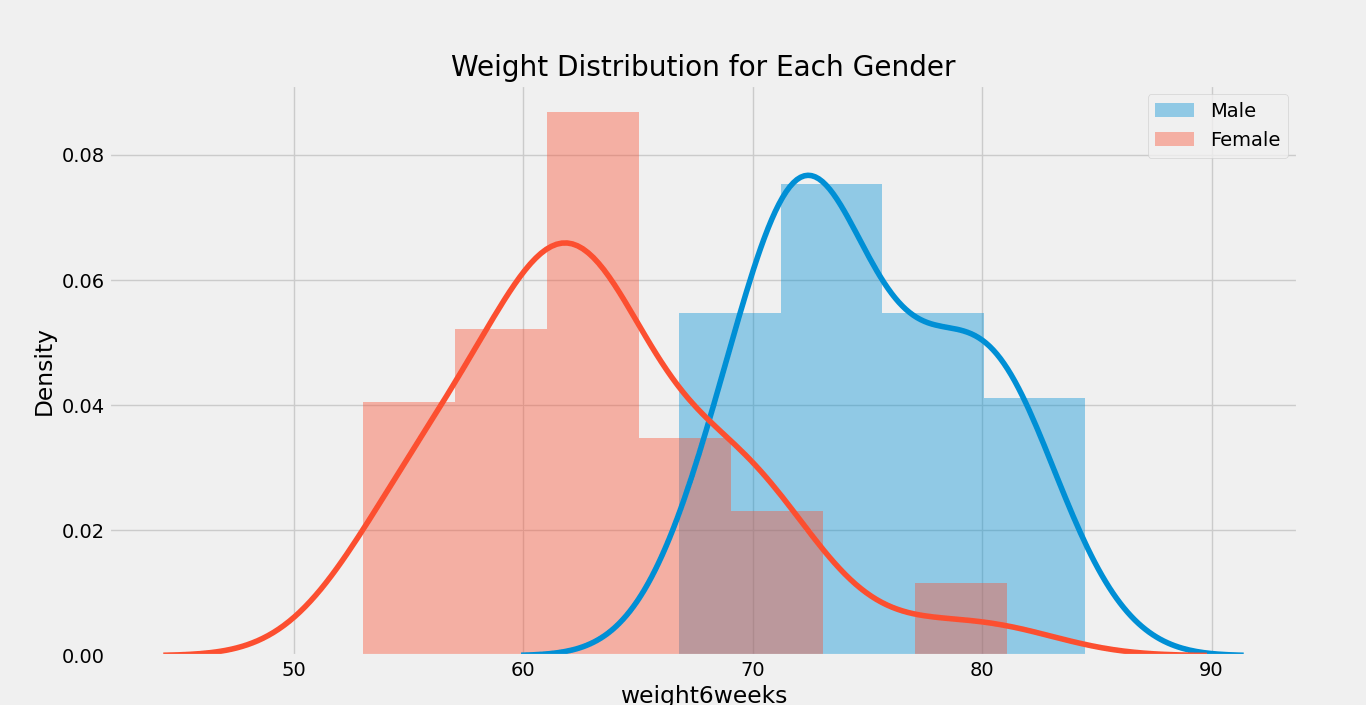
我们还可以为数据集中的每个性别绘制分布图。以下是相应的语法:
示例
f, ax = plt.subplots( figsize = (11,9) )
sns.distplot( mydata[mydata.gender == '1'].weight6weeks, ax = ax, label = 'Male')
sns.distplot( mydata[mydata.gender == '0'].weight6weeks, ax = ax, label = 'Female')
plt.title( 'Weight Distribution for Each Gender' )
plt.legend()
plt.show()
输出:
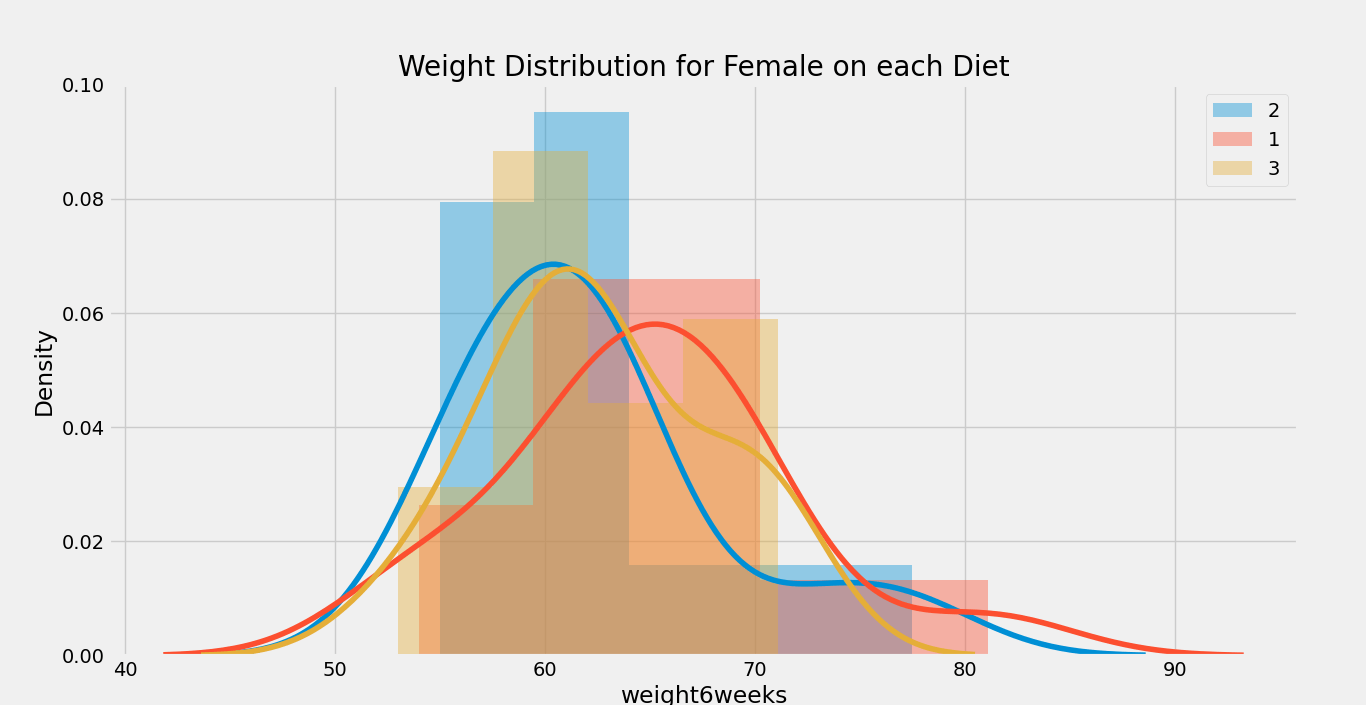
我们还可以使用以下函数来显示每个性别的分布图。
示例:
def infergender(x):
if x == '1':
return 'Male'
if x == '0':
return 'Female'
return 'Other'
def showdistribution(df, gender, column, group):
f, ax = plt.subplots( figsize = (11, 9) )
plt.title( 'Weight Distribution for {} on each {}'.format(gender, column) )
for groupmember in group:
sns.distplot(df[df[column] == groupmember].weight6weeks, label='{}'.format(groupmember))
plt.legend()
plt.show()
uniquediet = mydata.Diet.unique()
uniquegender = mydata.gender.unique()
for gender in uniquegender:
if gender != ' ':
showdistribution(mydata[mydata.gender == gender], infergender(gender), 'Diet', uniquediet)
输出:
图表 1:
图表2:

现在,我们将根据下面给出的代码片段计算’gender’列的均值、中位数、非零计数和标准差:
示例
print(mydata.groupby('gender').agg(
[ np.mean, np.median, np.count_nonzero, np.std ]
).weight6weeks)
输出:
mean median count_nonzero std
gender
81.500000 81.5 2.0 30.405592
0 63.223256 62.4 43.0 6.150874
1 75.015152 73.9 33.0 4.629398
正如我们所观察到的,我们已经根据性别估计了所需的统计测量。我们还可以根据性别和饮食对这些统计测量进行分类。
示例
print(mydata.groupby(['gender', 'Diet']).agg(
[np.mean, np.median, np.count_nonzero, np.std]
).weight6weeks)
输出:
mean median count_nonzero std
gender Diet
2 81.500000 81.50 2.0 30.405592
0 1 64.878571 64.50 14.0 6.877296
2 62.178571 61.15 14.0 6.274635
3 62.653333 61.80 15.0 5.370537
1 1 76.150000 75.75 10.0 5.439414
2 73.163636 72.70 11.0 3.818448
3 75.766667 76.35 12.0 4.434848
我们可以观察到饮食对女性的体重产生了轻微影响,但似乎对男性没有影响。
执行单因素方差分析检验
单因素方差分析的零假设为
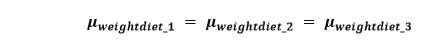
并且这个测试尝试检查这个假设是否成立。
我们首先要考虑确定置信水平为95%,这也意味着我们只接受5%的错误率。
示例
mymod = ols('Height ~ Diet', data = mydata[mydata.gender == '0']).fit()
# performing type 2 anova test
aovtable = sm.stats.anova_lm(mymod, typ = 2)
print('ANOVA table for Female')
print('----------------------')
print(aovtable)
print()
mod = ols('Height ~ Diet', data = mydata[mydata.gender=='1']).fit()
# performing type 2 anova test
aovtable = sm.stats.anova_lm(mymod, typ = 2)
print('ANOVA table for Male')
print('----------------------')
print(aovtable)
输出:
ANOVA table for Female
----------------------
sum_sq df F PR(>F)
Diet 559.680764 1.0 7.17969 0.010566
Residual 3196.086677 41.0 NaN NaN
ANOVA table for Male
----------------------
sum_sq df F PR(>F)
Diet 559.680764 1.0 7.17969 0.010566
Residual 3196.086677 41.0 NaN NaN
在上面的输出中,我们可以观察到两个p值(PR (> F)):男性和女性。 对于男性来说,我们不能在95%的置信水平下接受零假设,因为p值大于alpha值,即0.05 < 0.512784。因此,在提供这三种类型的饮食后,男性的体重没有发现差异。 对于女性来说,由于p值PR (> F)低于错误率,即0.05 > 0.010566,我们可以拒绝零假设。这个陈述表明,我们对于女性在饮食方面的体重差异非常有信心。 因此,现在我们了解了饮食对女性的影响;然而,我们不清楚不同饮食之间的差异。因此,我们将利用图基HSD(Honest Significant Difference)测试进行事后分析。 让我们考虑以下代码片段。示例
from statsmodels.stats.multicomp import pairwise_tukeyhsd, MultiComparison
# using the female data only
mydf = mydata[mydata.gender == '0']
# comparing the height between each diet, using 95% confidence interval
multiComp = MultiComparison(mydf['Height'], mydf['Diet'])
tukeyres = multiComp.tukeyhsd(alpha = 0.05)
print(tukeyres)
print('Unique diet groups: ', multiComp.groupsunique)
输出:
Multiple Comparison of Means - Tukey HSD, FWER=0.05
=====================================================
group1 group2 meandiff p-adj lower upper reject
-----------------------------------------------------
1 2 -3.5714 0.5437 -11.7861 4.6432 False
1 3 -8.7714 0.0307 -16.848 -0.6948 True
2 3 -5.2 0.2719 -13.2766 2.8766 False
-----------------------------------------------------
Unique diet groups: [1 2 3]
如上所示,我们可以观察到,我们只能在第一种和第三种类型的饮食中拒绝零假设,这意味着饮食1和饮食3的体重存在统计学上的显著差异。
 极客笔记
极客笔记