Python Biopython模块
大多数人都思考过为什么Python与其他编程语言相比增长如此迅速?是的,Python确实在很短的时间内变得非常有名,现在我们可以在每个领域看到Python的应用。而且,是的,每个领域意味着技术可以被看到的每个领域。Python编程语言不仅仅限于编程或开发目的,现在我们还可以在医学、商业、国防、电子商务等各个领域看到其应用。Python取得巨大发展和影响的主要原因是其简单性和众多附带的库。许多领域之所以取得新的成功和发展,正是因为在其中使用了Python。
如果我们再次看一下我们提到的领域的名称,我们会发现我们还提到了医学领域。现在,我们中的许多人会想到Python如何在医学领域中有所帮助。对于这个问题的答案并不仅限于医院或诊所中使用的医疗设备,Python还用于医学的各个其他领域。Python可以在医学信息学领域中发挥作用,而我们在这里不必混淆遗传学(生物技术)和生物信息学。
注意:生物信息学是一个跨学科领域,涉及生物学和计算机科学、数学、物理等各个领域的研究。
在Python中,我们有一个非常著名的模块,即Biopython,用于生物信息学,许多科学家现在正使用此模块进行研究,因此该模块的使用正在迅速增加。在本教程中,我们将学习这个Biopython模块,并稍微了解一下它。我们还将通过一个示例学习它的安装以及在生物信息学研究中如何使用它。
Biopython模块
在Python甚至大多数编程语言中,Biopython不仅是最受欢迎的,也是最大的生物信息学包。Biopython模块包含许多不同的子包,用于执行常见的生物信息学任务。Biopython模块主要是用Python编写的,但也包含C代码,由Chang和Chapman开发。Biopython包中的C代码用于优化模块的复杂计算部分。Biopython可以在多个操作系统上运行,如Windows、Linux、UNIX、Mac OS X等。
在我们开始学习Biopython模块之前,我们必须对生物信息学术语(如DNA、RNA、蛋白质序列、基因组序列等)有一个基本的了解。否则,将很难理解该模块的工作和功能。除了生物信息学的基本术语外,我们还应确保我们的系统中存在最新版本的Python,并且我们熟悉pip安装程序。
Biopython模块:介绍
Biopython模块是一个由不同Python模块组成的集合,提供了处理DNA、RNA和蛋白质序列等各种遗传结构的许多不同功能。在这里我们提到的蛋白质序列操作可以是在蛋白质序列中找到子序列,反向补充DNA序列等。在Biopython模块中,我们提供了许多解析器,并且借助这些解析器,我们可以读取所有主要的遗传数据库,比如SwissProt、GenBank、FASTA等等。这些解析器也非常有助于读取主要的包装器/接口,这些接口在运行其他流行的生物信息学工具/软件(如Entrez、NCBI BLASTN等)时非常有帮助。借助Biopython模块,我们可以使用Python编写的Python程序在Python环境中完成所有这些操作。
Biopython模块:特点
到目前为止,我们肯定已经了解到Biopython模块的重要性以及它对所有与生物信息学领域相关的人员的帮助。现在,我们将讨论Biopython模块提供的功能以及其著名之处。以下是Biopython模块的一些重要特点:
- Biopython模块具有易于学习、易于移植和非常清晰的语法。
- Biopython模块为我们提供了处理蛋白质序列格式的选项。
- Biopython模块为我们提供了各种工具,以管理不同类型的蛋白质结构。
- Biopython模块是面向对象的、解释性的和交互式的,与Python相同。
- Biopython模块为我们提供了访问许多本地生物信息学服务的途径,包括Clustalw、Blast、EMBOSS等。
- Biopython模块支持各种生物信息学文件格式,如SCOP、FASTA、Medline或PubMed、PDB、ExPaSY相关格式和GenBank。
- 借助Biopython模块,我们可以访问各种在线服务和数据库,如NCBI服务,包括PubMed、Blast、Entrez等,以及ExPaSY服务,如Prosite和SwissProt。
- Biopython模块还提供了BioSQL的选项,这是一组标准的SQL表,我们可以使用它们来存储序列、特征和注释。
我们现在已经看到了Biopython模块的所有重要特点,现在我们可以理解这个模块在生物信息学领域的所有工作中是多么有用。
Biopython模块:目标
众所周知,Biopython模块是生物信息学领域所有实地工作和研究工作的最佳Python包,但它在构建时肯定有一些目标。一般来说,Biopython模块的目标是通过Python语言提供对生物信息学工作所需的所有数据和工具的标准、简单和广泛的访问。但这并不是构建这个模块的唯一目标;还有其他主要目标。我们将讨论Biopython模块建立的所有这些主要目标,并将它们列出在本节中。
以下是构建Biopython模块的所有主要或具体目标的列表:
- Biopython模块旨在帮助进行基因组数据分析。
- Biopython模块旨在提供高质量和可重用的脚本和模块。
- 构建Biopython模块的目标之一是提供对所有生物信息学资源的标准化访问。
- Biopython模块还旨在具有快速数组操作,可用于PDB、Markov模型、NaiveBayes和聚类代码。
所以,这些都是Biopython模块构建和引入Python作为生物信息学软件包的特定和主要目标。
Biopython模块:优点
现在我们已经了解了Biopython模块的特点以及如何对所有与生物信息学领域有关的人都非常有帮助。我们可以很容易地描述出该模块的一些优点,但是仍然有一些优点是我们无法从列出的特点或目标中猜测出来的。因此,在本节中,我们将看到Biopython模块的所有优点以及它在许多方面的帮助。
以下是使用Biopython模块进行生物信息学研究和工作的一些优点:
- Biopython模块为我们提供了在聚类过程中使用的不同微阵列数据类型。
- Biopython模块还为医学应用中使用的所有类型的日志数据提供支持。
- Biopython模块为我们提供了基于教程风格的清晰文档。
- Biopython模块在阅读和写入具有树状视图的不同文件时非常有帮助。
- Biopython模块非常有用,因为它通过提供各种子模块来支持解析生物信息学文件为通用序列加特征类或格式特定记录对象的解析器开发。
- Biopython模块还支持用于PDB表示、分析和解析的结构数据。
- Biopython模块还为各种生物信息学数据库提供支持,例如BioSQL数据库(在生物信息学项目中广泛使用的标准数据库)。
所以,这是我们使用Biopython模块时拥有的所有优点的列表,它也揭示了这个模块对于与生物信息学领域有关的每个人来说非常有帮助和有用。
Biopython模块:安装
现在,我们将学习如何在Python程序中实现和使用Biopython模块。我们首先需要在系统中安装Biopython模块,然后才能在Python程序中导入和使用该模块的功能。因此,我们将在这里学习Biopython模块在系统中的安装过程,并检查我们设备上安装的Python的兼容性。这是因为Biopython模块支持Python 2.5以上的版本,而低于2.5的版本不支持安装和导入Biopython模块。因此,我们首先必须确保系统中安装的Python是符合要求或最新版本的。
现在,如果我们不知道系统中安装的Python版本,并且想要检查它,那么我们可以在命令提示符终端中使用以下命令:
python --version
当我们按下回车键后,我们系统中安装的Python版本将会显示出来,就像我们在输出图片中看到的那样。
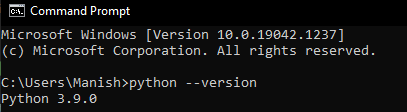
在显示的版本中,可以看到系统中安装的Python版本高于所需版本。但是,如果系统中的Python版本与所需版本2.5不相等或不高于,则我们应该先更新它,然后才能继续安装部分。
注意:有许多其他方法可以检查系统中安装的Python版本,但我们将首选此方法,因为这是最简单最简单的方法。
现在,在检查系统中安装的Python版本后,我们将继续安装Biopython模块,并使用pip安装程序来安装此模块。我们将在命令提示符终端中使用以下pip安装程序命令来在系统中安装Biopython模块:
pip install biopython
当我们在命令后按下回车键后,pip安装程序将开始在我们的系统中安装Biopython模块。
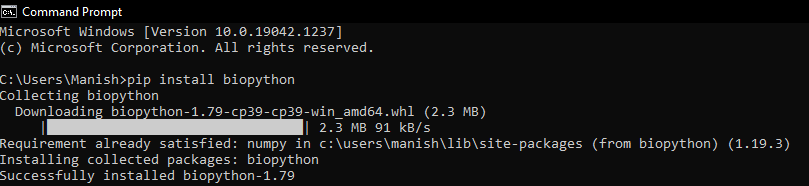
Biopython Module 现已成功安装在我们的系统中,现在我们可以将其导入到 Python 程序中,以使用其功能并学习其实现。
Biopython 模块:实现
为了了解 Biopython 模块的工作方式以及它如何帮助解析生物信息学文件,我们首先要创建一个示例FASTA文件(这里 ‘fasta’ 是指来自生物信息学软件的文件格式序列)。在 FASTA 文件格式中,文件中的序列按顺序排列,每个序列在文件中都有自己的ID、名称、描述和实际序列数据。
我们首先要打开系统中的记事本,并在其中写下以下内容:

现在,我们需要将这个记事本文件保存为名为’SampleFile1.fasta’的文件,并且我们需要将其保存在Python安装的同一目录中,这样我们在打开文件时就不需要写整个目录。现在我们将使用Biopython模块在Python程序中,并通过解析我们创建的示例fasta文件来学习其实现。
请看下面的Python程序,在这里我们使用Biopython模块的函数解析了示例fasta文件:
# Importing required functions from Biopython module
from Bio.SeqIO import parse
from Bio.SeqRecord import SeqRecord
from Bio.Seq import Seq
# Open the sample FASTA file we have created
sampleFile = open("SampleFile1.fasta")
# Parsing the file in the Python program
parseRecords = parse(sampleFile, "fasta")
# Using for loop to printing attributes of files
for record in parseRecords:
# Printing multiple attributes of the file
print("Id of FASTA File: %s" % record.id)
print("Name of FASTA File: %s" % record.name)
print("Description of FASTA File: %s" % record.description)
print("Annotations in FASTA File: %s" % record.annotations)
print("Sequence Data in FASTA File: %s" % record.seq)
输出:
Id of FASTA File: sampleFile|P2426|FMS1_ECOLI
Name of FASTA File: sampleFile|P2426|FMS1_ECOLI
Description of FASTA File: sampleFile|P2426|FMS1_ECOLI CS1 is a fimbrial subunit of the precursor (Have CS1 pilin)
Annotations in FASTA File: {}
Sequence Data in FASTA File: MKLKKTIGADALATLFATMGASAVEKTISVTASVDMTVDLLQSDGSALPNSVALTYSPAVNNFEAHTINTVVQTNDSDKGVVVKLSAMPVLSNVLNPTLQIPVSVNFAGKPLSTTGITIDSNDLNFASSGVNKVSMTQKLSIHADATRVTGGALTAGQYQGLVSIILTKSTTTTTTTKGT
Id of FASTA File: sampleFile|P2631|FMS3_ECOLI
Name of FASTA File: sampleFile|P2631|FMS3_ECOLI
Description of FASTA File: sampleFile|P2631|FMS3_ECOLI CS3 is a fimbrial subunit of the precursor (Have CS3 pilin)
Annotations in FASTA File: {}
Sequence Data in FASTA File: MLKIKYLLIGLSKSAMSSYSLAAAGPTLTKELALTVLSPAALDATWAPQDNLTLSNTGVSNTLVGVLTLSNTSIDTVSIANTNVSDTSKNGTVTFAHETNNSASFATTISTDNANITLDKNAGNTIVKTTNGSPLPTNLPLKFITTEGNEHLVSGNYRANITITSTIKGGGTKKGTTDKK
解释:
首先,在程序中我们使用“from”关键字导入了Biopython模块的不同工具,如parse、SeqRecord和Seq。然后,我们使用open()函数打开了我们在程序中创建的样本fasta文件。然后,我们对我们初始化打开样本文件的变量,即sampleFile,使用parse()函数。然后,我们循环遍历parseRecords变量(文件解析的初始化变量)来打印文件中的不同属性和属性。
我们显示了以下属性及其对应的Biopython模块的函数:
- 我们使用record.id打印了文件中的id,
- 我们使用record.name打印了文件中序列的名称,
- 我们使用record.description打印了文件中序列的描述,
- 我们使用record.annotations打印了序列的注释,
- 最后,我们使用record.seq打印了样本文件中的序列。
正如我们在输出中所看到的,样本fasta文件的所有属性都被成功打印出来了,并且这些属性首先被打印出来的是第一个序列,然后是第二个序列。
这是一个使用Biopython模块进行生物信息学工作并使用Python程序解析生物信息学软件文件的示例文件示例。
 极客笔记
极客笔记